
ENZYMY , KT╙RE TNí DNA NA FRAGMENTY I úíCZí JE POZWALAJíC NA NIEZLICZONí ILOª╞ KOMBINACJI TYCH»E.
RESTRYKTAZY - tn▒ DNA tworz▒c powtarzalny komplet fragment≤w.
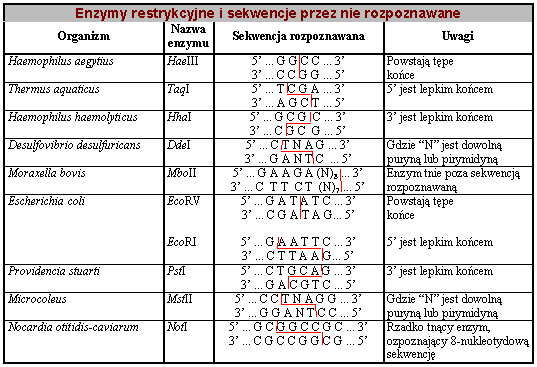
Restryktazy (inaczej enzymy restrykcyjne , endonukleazy restrykcyjne) - to enzymy izolowane z bakterii , zdolne do rozpoznawania specyficznych sekwencji w DNA (z regu│y s▒ to sekwencje palindromowe) i do przecinania dwuniciowej cz▒steczki DNA w ╢ci╢le okre╢lonym miejscu , w obrΩbie lub okolicy sekwencji rozpoznawanej. Otrzymywane fragmenty DNA nie s▒ losowe a w ka┐dym pr▒┐ku na ┐elu znajduj▒ siΩ cz▒steczki DNA o identycznej sekwencji nukleotydowej. Z regu│y r≤┐ne enzymy rozpoznaj▒ odmienne sekwencje DNA. Istniej▒ jednak wyj▒tki - tzw. izoschizomery - enzymy izolowane z r≤┐nych organizm≤w ale rozpoznaj▒ce te same sekwencje. Zdarza siΩ tak┐e , ┐e dwa enzymy wytwarzaj▒ takie same lepkie ko±ce , mimo rozpoznawania r≤┐nych sekwencji DNA. Umo┐liwia to klonowanie DNA strawionego jednym enzymem w wektorze strawionym innym , daj▒cym takie same lepkie ko±ce.
Nazewnictwo opiera siΩ na literowych skr≤tach , w kt≤rych pierwsza litera pochodzi od rodzaju bakterii , a druga i trzecia od gatunku. NastΩpna litera oznacza szczep lub lub typ a kolejne enzymy z danego szczepu lub typu otrzymuj▒ litery rzymskie.
Jednostka enzymu restrykcyjnego to taka jego ilo╢µ , kt≤ra trawi kompletnie 1mg DNA faga l (oko│o 50 kb) w czasie 1 godz. w temperaturze 37░C.
Podzia│ restryktaz wed│ug rodzaju wytwarzanych ko±c≤w :
- tΩpe ko±ce - nici rozciΩte naprzeciwko siebie - wszystkie nukleotydy s▒ sparowane z komplementarnymi nukleotydami na przeciwnym │a±cuchu
- lepkie ko±ce - 3` lub 5` ssDNA (jednoniciowe) ogony na obu ko±cach utworzone przez niesymetryczne ciΩcie , komplementarne do podobnych tworzonych w innych cz▒steczkach DNA przez te same enzymy restrykcyjne niezale┐nie od ╝r≤d│a DNA , co pozwala na ligowanie DNA nawet bardzo r≤┐ni▒cych siΩ gatunk≤w czyli formowanie chimerycznych moleku│.
Modyfikacje ko±c≤w :
- tΩpych , poprzez do│▒czenie adaptor≤w (kr≤tkich fragment≤w DNA zako±czonych z jednej strony na tΩpo z drugiej na lepko , specyficznie dla danej restryktazy) lub linker≤w (kr≤tkich fragment≤w DNA zawieraj▒cych okre╢lone miejsce restrykcyjne , zako±czonych na tΩpo) , przed do│▒czeniem tych ostatnich nale┐y zmetylowaµ genomowy DNA odpowiedni▒ metylaz▒ a po ligacji strawiµ DNA dan▒ restryktaz▒ uzyskuj▒c lepkie ko±ce
- lepkich , kt≤re maj▒ cofniΩt▒ niµ 3` Mo┐na je wype│niµ przy pomocy fragmentu Klenowa polimerazy DNA E.coli i kompletu nukleotyd≤w.
- lepkich , kt≤re maj▒ cofniΩt▒ niµ 5` Mo┐na je wytΩpiµ obcinaj▒c wysuniΩty jednoniciowy odcinek DNA przy u┐yciu nukleazy S1 lub polimerazy I E.coli wykorzystuj▒c jej aktywno╢µ egzonukleolityczn▒ 3`«5`.
Podzia│ restryktaz wed│ug sekwencji rozpoznawanej :
- czw≤rkowe , rozpoznaj▒ sekwencjΩ DNA z│o┐on▒ z czterech nukleotyd≤w. Statystycznie w dowolnym DNA takich miejsc jest du┐o - co 256 bp. Restryktazy takie mog▒ strawiµ DNA na bardzo ma│e kawa│ki.
- sz≤stkowe , rozpoznaj▒ sekwencjΩ DNA z│o┐on▒ z sze╢ciu nukleotyd≤w. Dowolne miejsce restrykcyjne z│o┐one z sze╢ciu nukleotyd≤w wystΩpuje statystycznie co oko│o 4096 bp w DNA , w kt≤rym ilo╢ci poszczeg≤lnych nukleotyd≤w s▒ r≤wne. Proporcje te zmieniaj▒ siΩ w zale┐no╢ci od organizmu , dlatego dob≤r enzymu sz≤stkowego i warunki nale┐y ustaliµ eksperymentalnie.
- ≤semkowe , stosowane niezbyt czΩsto. Tn▒ DNA bardzo rzadko.
Zastosowania :
- Konstrukcja map restrykcyjnych. Analizuj▒c na ┐elach produkty trawienia danego DNA r≤┐nymi enzymami restrykcyjnymi , stosowanymi pojedynczo , w kombinacjach oraz w ilo╢ciach wystarczaj▒cych lub niewystarczaj▒cych do pe│nego strawienia preparatu , mo┐na ustaliµ wzajemne po│o┐enie i odleg│o╢ci pomiΩdzy sekwencjami rozpoznawanymi przez te enzymy. Mapa restrykcyjna to obraz cz▒steczki DNA , na kt≤rym zaznaczone s▒ miejsca rozpoznawane przez r≤┐ne enzymy restrykcyjne z uwzglΩdnieniem odleg│o╢ci miΩdzy nimi wyra┐onej w nukleotydach.
- Klonowanie i obr≤bka DNA. DNA pociΩty enzymami restrykcyjnymi mo┐na poddawaµ ligacji z wektorem. W ten spos≤b tworzone s▒ zrekombinowane plazmidy czyli plazmidy zawieraj▒ce sklonowany DNA , kt≤ry mo┐na p≤╝niej poddawaµ r≤┐nym manipulacjom przy u┐yciu odpowiednich enzym≤w restrykcyjnych.
- Badanie polimorfizmu miejsc restrykcyjnych (RFLP).
Metody alternatywne fragmentowania DNA - mechaniczna fragmentacja DNA (bardzo rzadko stosowane):
- Shearing przy u┐yciu strzykawki z ig│▒. Stopie± fragmentacji zale┐y od grubo╢ci ig│y i ilo╢ci przepuszcze±. Uzyskuje siΩ fragment zako±czone na tΩpo poniewa┐ nici DNA pΩkaj▒ zwykle naprzeciwko siebie. R≤┐norodno╢µ powsta│ych fragment≤w jest bardzo du┐a gdy┐ pΩkniΩcie mo┐e nast▒piµ w dowolnym miejscu.
- Sonikacja. Preparat DNA poddaje siΩ dzia│aniu ultrad╝wiΩk≤w. Powoduj▒ one pΩkanie nici. Odpowiedni stopie± fragmentacji mo┐na uzyskaµ dobieraj▒c eksperymentalnie warunki i czas sonikacji.
LIGAZY - trwale │▒cz▒ pociΩte fragmenty.
.gif)
Ligazy DNA katalizuj▒ formowanie wi▒za± fosfodiestrowych pomiΩdzy ko±cem hydroksylowym 3` a ko±cem fosforowym 5` DNA. Pozwala to na reperowanie jednoniciowych przerw w dupleksie DNA , │▒czenie fragment≤w restrykcyjnych posiadaj▒cych homologiczne lepkie czy te┐ nawet tΩpe ko±ce.
Dwa najczΩ╢ciej stosowane enzymy to ligaza DNA E.coli i ligaza DNA z faga T4. Tylko ta ostatnia jest w stanie wydajnie │▒czyµ tΩpe ko±ce , nawet w normalnych warunkach reakcji. Wad▒ jest jej mniejsza specyficzno╢µ rozpoznawania struktury ko±c≤w , co daje wiΩksze t│o w do╢wiadczeniach spowodowane nieprawid│owymi ligacjami.
 BIBLIOTEKI A MO»E BANKI DNA I CO SI╩ W NICH PRZECHOWUJE.
BIBLIOTEKI A MO»E BANKI DNA I CO SI╩ W NICH PRZECHOWUJE.
WEKTOR PO úACINIE ZNACZY PRZEWO¼NIK.


⌐ 1997, 1998 Biologia Molekularna w Internecie Webmaster





